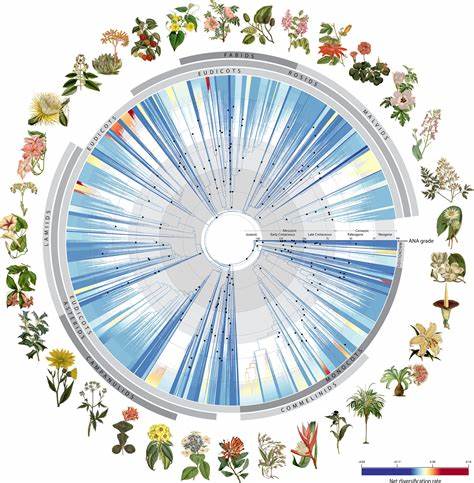
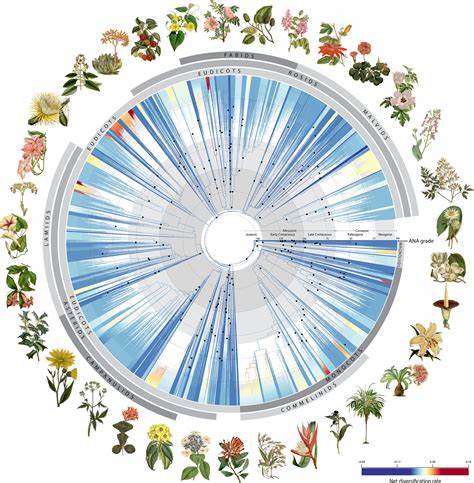
随着生物多样性研究的深入,科学家们对物种鉴定技术的需求日益增长。传统的DNA条形码方法依赖于特定的短序列片段,在实践中尽管带来了重要突破,但面对整个生命树的多样性,存在普遍适用性不足和识别分辨率有限的难题。最近,一项由布鲁诺·A·S·德·米德罗斯等科学家提出的创新方法,变革了这一领域。他们开发了名为varKoding的技术,利用极低覆盖度的基因组“浅序列”数据将复杂的基因组信息转化为二维图像,实现对物种的精准识别。这种基于机器学习的图像识别模型提供了超越传统方法的新路径,促进了生命树上不同生物门类的统一识别。传统DNA条形码多局限于特定分类单元,如以动物线粒体COI基因为主的动物条形码,或是植物中多为叶绿体基因片段的条形码。
这些短序列在不同物种间序列相似度较高时易导致鉴定错误,且无法涵盖所有生物类群。加之PCR扩增依赖特异性引物,面对基因组结构复杂或DNA降解严重的样品时常遭遇失败。相较之下,varKoding创新地将基因组序列中的k-mer(长度为k的短序列)频率信息映射为图像,利用深度神经网络,特别是视觉变换器(ViT)架构,在图像分类任务上表现卓越,实现低覆盖度数据下的高识别精度。其优势不仅仅是无需组装复杂基因组,避免了数据处理的庞大资源消耗,还能减轻对扩增引物和目标序列的依赖,使得历史标本、降解DNA样本甚至环境DNA样本的分析成为可能。主要研究团队采用涵盖多物种、多分类单元的植物与多样生物数据集进行系统测试。在Malpighiales植物类群中,varKoding能够在极低(远低于1×)的基因组覆盖度条件下实现90%以上的准确率,超越了Skmer等基于k-mer的传统方法,以及常规的条形码策略。
在跨界应用中,无论是昆虫、真菌还是细菌类群,varKoding均展示了良好的识别能力,显示其在整个生命树上的普适性。varKoding的核心思想是利用k-mer频率的秩变换(ranking),减少高频异常k-mer的干扰,并结合t-SNE等降维技术优化二维映射,从而生成具有物种特异性的基因组“签名”图像。神经网络能够捕捉这些图像中微妙而复杂的特征差异,实现分类鉴定。训练过程中,通过数据增强技术如CutMix和MixUp提升模型泛化能力,减少对大规模标记数据的依赖,证实即使样本量较小仍能获得高准确率。此外,研究中提出的多标签分类策略有效应对污染与数据质量差异造成的不确定性,避免错误的强制分类,提升模型的实用价值。另一个重要特点是计算效率。
传统的基于基因组比对或繁复组装的鉴定方法随着样本数量增加呈现指数级时间和存储增长,而varKoding的模型大小固定,训练时间随数据线性增加,便于扩展至全基因组规模和全球物种数据库。结合如今高通量测序及便携测序设备的快速数据生成能力,varKoding支持野外即时鉴定及法医、生物监测等实际应用,推动基因组学向实时化和多样化方向迈进。普遍应用varKoding能够大幅加速自然历史博物馆中珍贵标本的数字化识别,加快物种发现和鉴定流程,促进生态系统保护和资源管理。面对环境DNA采样带来的混合样品问题,预计未来结合单分子长读长序列技术,varKoding有望分析混合样品中的多重物种成分,实现复杂微生物组和生态环境的高通量监测。整体而言,varKoding代表了DNA鉴定技术从传统基因序列比对向智慧图像识别与机器学习融合发展的新范式。它打破了条形码方法的单一基因局限,以极少的基因组数据实现跨界生物类群的高精度鉴定,显示出强大的普适性和实用性。
借助公共数据库如NCBI序列阅读档案的大规模数据积累,结合先进的神经网络技术和算法优化,varKoding为探索地球生命多样性注入了强劲动力。在未来,配合全球物种基因组测序计划和便携式测序技术的普及,varKoding必将推动生物学、生态学、环境科学和法医鉴定等多领域的革命性进步。生物多样性的深度揭示和持续监控将为应对气候变化、物种保护和可持续发展提供科学基础。随着技术的成熟与推广,varKoding有望成为各类生物识别任务的新标准工具,推动生命科学进入一个以数据智能驱动的新时代。