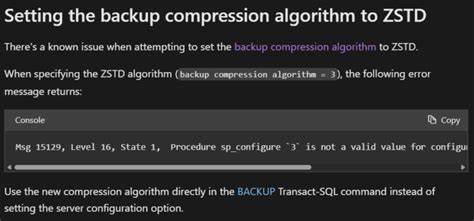
随着基因组测序技术的飞速发展,海量的基因组数据以文本格式存储,特别是FASTA格式在生物信息学中被广泛使用。然而,对于这些庞大的文本文件,如何高效压缩以节省存储空间和传输带宽,成为科研和企业领域日益关注的热点。近年来,Zstandard(简称ZSTD)压缩算法因其高速压缩能力和较优的压缩比而备受青睐。最新研究表明,将FASTA文件中的换行符去除,能够显著提高ZSTD的压缩效率,甚至可以实现压缩比提升十倍之多。本文将深入解析为何换行符对压缩率产生如此重大影响,如何操作以最大化压缩效果,以及ZSTD长距离匹配模式在基因组数据压缩中的实际应用和优势。FASTA文件是一种基因组序列的文本存储格式,通常每行包含固定长度的碱基序列,行尾以换行符(0x0A)结束。
这些换行符虽然方便人工阅读和序列查看,但在压缩角度来看,并不承载实际生物信息,被归类为非语义的"美容"字符。这正是影响压缩性能的关键所在。ZSTD压缩算法依赖于上下文窗口内的重复匹配,尤其是在长距离匹配(long range match)模式下,能够识别文件中遥远重复片段,实现更深度的重复消除。由于FASTA文件中每60个碱基就插入一个换行符,这些频繁出现的换行符序列会破坏相邻重复序列在哈希表中的匹配,造成长距离匹配的断裂,大幅降低了压缩效果。为了验证这个假设,研究者们使用了包含661405个细菌基因组组装体的经典大数据集,总大小约2.46TB。使用默认参数的Zstandard压缩该数据集获得了约3倍的压缩比,压缩后的文件大小约为777GB。
启用长距离匹配功能(--long)稍微提升了压缩比至3.8倍,大小降至641GB,但距离专门的基因组压缩器还有相当差距。随后,将FASTA文件中的换行符完全移除(使用seqtk seq -l 0工具),即可获得一份完全连续的碱基序列文本。对处理后的该文件再次使用Zstandard的长距离匹配模式,压缩比飞速提升至11倍,压缩后文件大小缩小为232GB,且压缩时间仅增加约20%。进一步将ZSTD的窗口大小增加到最大允许的2GB(--long=31参数),压缩比更是飙升至31倍,文件缩小至80GB,压缩时间约增加80%。这些结果表明,去除换行符可以大幅降低序列碎片化对重复匹配的破坏,为ZSTD算法提供了更完整的上下文信息,从而提升匹配成功率和压缩效率。与此同时,需要注意的是使用超大窗口参数(--long=31)会导致解压时必须传入相同参数,降低了一定程度的兼容性。
尽管如此,考虑到Zstandard压缩速度快且支持流式压缩,这种方案在大规模基因组数据处理流程中极具实际应用价值。相比传统通用压缩工具如gzip,ZSTD不仅压缩速度快出数倍,还能通过优化输入文件结构大幅提升压缩比。而与专门的基因组压缩工具如MiniPhy相比,虽然压缩比仍有差距,但ZSTD提供了一种平衡速度与效率的可行路径。深入理解FASTA文件格式和压缩算法之间的交互,对于基因组数据存储和传输系统的设计至关重要。换行符作为视觉便利的排版成分,其在压缩上下文中却属于累赘,去除或减少其频率是优化流程中不可忽视的一步。未来,结合专用的预处理步骤和高效的长距离匹配算法,有望实现更高效的基因组压缩框架。
同时,相关社区实践也表明,整合压缩时的预处理策略,诸如去换行、过滤空白字符等,对于提升多种压缩工具的性能均适用。总结而言,通过去除FASTA文件中的换行符,充分利用Zstandard的长距离匹配功能,能够实现压缩效率上质的飞跃。该方法不仅极大节约存储空间,还提高了数据传输及处理效率,为海量基因组数据的存储和分析提供了切实可行的技术方案。研究和应用者应重视输入文件的格式规范化,以发挥压缩算法的最大潜力。随着基因数据规模的持继扩大,类似的细节优化将在未来数据管理体系中扮演举足轻重的角色。 。