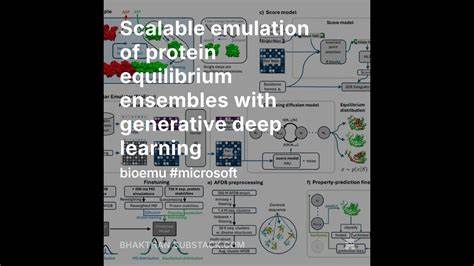
蛋白质作为生命的基本功能单元,其结构和动态行为直接关乎细胞的各种生物学过程与疾病的发生机制。理解蛋白质如何在溶液中以多种构象存在,揭示其构象间的转变,对于药物设计和生物技术开发具有重要意义。然而,蛋白质的功能往往依赖于其复杂的构象变化,这些变化通常涉及从主状态到稀有或过渡态的多种长寿命构象。传统的分子动力学模拟虽然能够提供精细的动力学细节,但由于涉及极大的计算资源和长时间尺度,限制了其在大规模蛋白质功能预测中的广泛应用。 随着深度学习技术的迅猛发展,科研人员开始尝试借助人工智能模型来模拟蛋白质的平衡构象组,以期突破传统模拟的效率瓶颈。生成式深度学习便在此背景下应运而生,提供了一种全新的蛋白质结构动态模拟思路。
通过训练神经网络模型来学习蛋白质序列、静态结构数据及分子动力学模拟结果,实现对蛋白质构象空间的高效采样,生成大量符合平衡态分布的独立构象。此方法不仅大幅度缩短了计算时间,还能保持与实验数据的高度一致性,极大促进了蛋白质功能研究的可扩展性和实用性。 BioEmu便是该领域的一个典型代表,它融合了AlphaFold的序列-结构编码优势和基于扩散模型的三维结构生成能力。通过跨越三个训练阶段:首先在经过处理的AlphaFold数据库上预训练以激励模型能为单一序列生成多样结构;随后利用总时长超过200毫秒的分子动力学模拟数据进行深入训练;最后结合超过五十万条实验测得的蛋白质稳定性数据进行细化,该体系精准地捕获了蛋白质构象的热力学特性与功能相关的动态机制。 BioEmu展现了强大的泛化能力和高度准确度。它能够成功预测包括大域运动、局部解折叠以及隐匿结合口袋的形成等多样的构象变化,采样成功率介于55%到90%之间。
此外,在模拟蛋白质的折叠平衡与天然状态构象转变时,模型的自由能差误差低于1千卡/摩尔,并实现了比传统分子动力学模拟高出四到五个数量级的加速。在利用生成的平衡构象组直接预测蛋白质的稳定性及突变体效应时,也达到了高相关系数和低误差,表现出较强的生物物理学解释能力。 生成式深度学习模型的优势不仅体现在速度和精度上,更为蛋白质设计和药物发现开辟了广阔的前景。快速生成的构象集允许研究者高效筛查可能的结合口袋和变构位点,推进对蛋白质功能机制的理解。此外,该方法的训练策略证明,通过集成大量多样性的模拟与实验数据,模型的预测误差会显著下降,显示出随着训练数据丰富深入,蛋白质功能预测的规模化和准确化是完全可行的。 面对蛋白质内在的复杂动态,单一结构预测往往难以准确反映其真实生理状态。
生成式深度学习通过直接建模蛋白质的构象分布,切实捕捉了其在生理环境中可能出现的多种功能相关构象。此方法促进了蛋白质组级别功能动力学的研究,有望推动精准药物设计和生物工程技术的创新,为理解复杂生物系统提供新的计算工具。 当然,蛋白质研究领域仍存在诸多挑战。蛋白质的功能不仅依赖结构本身,更受溶液环境、蛋白质组分及其相互作用的调控。当前生成模型主要基于理想或有限环境下的数据,如何融入更为复杂的生物化学和生物物理条件,是未来研究的重要方向。同时,如何确保生成的构象组真正代表生物体内的平衡状态,避免过拟合或模型偏差,也是需要持续关注的问题。
展望未来,生成式深度学习与高性能计算、实验数据的深度整合将不断推动蛋白质结构生物学研究的前沿。随着数据的不断累积和算法的不断优化,能够高速准确地模拟蛋白质平衡构象组的人工智能平台将成为生命科学领域的核心工具。它不仅能极大降低分子动力学模拟对计算资源的需求,还能加快新颖蛋白质功能的发现与设计,助力精准医疗和生物技术的飞跃发展。最终,结合多模态数据源,挖掘蛋白质结构动态与功能的深层联系,将实现对生命分子机器前所未有的理解和掌控。 。