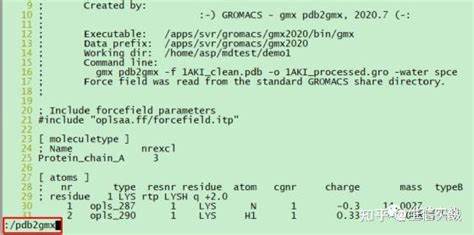
在分子模拟领域,GROMACS作为广泛使用的分子动力学模拟软件,以其高效和强大的功能深受科研人员喜爱。计算分子中的残基间距离是理解蛋白质结构变化、分子互作及动力学行为的重要分析手段。本文将详细介绍如何在GROMACS中准确计算残基间距离,涵盖操作流程、关键选项解释、常见问题及实际应用案例,帮助用户深入掌握此项分析技能。 首先需要明确计算残基间距离的意义。残基是蛋白质结构的基本组成单位,分子动力学模拟中的距离变化反映了分子构象的动态特征及功能相关变化。通过精确测量特定残基之间的距离,可以揭示可能的结合位点、构象转变过程以及蛋白质 - 配体相互作用等信息。
GROMACS中计算残基间距离主要依靠distance工具(gmx distance)。此工具可以针对模拟轨迹中的原子或分子集合,计算它们之间的距离随时间的变化。使用前需要准备好模拟生成的轨迹文件(如.xtc、.trr格式)和对应的结构文件(如.gro、.pdb格式)。 启动计算前,用户需指定距离计算的对象,通常是通过索引文件选定特定残基的原子。通过make_ndx命令生成自定义的索引组,可以将残基中特定原子(例如CA原子)划分为单独组别。索引文件的制定是计算残基间距离的关键步骤,保证所选择的残基或原子正确无误。
执行gmx distance命令时,系统会提示选择两组原子/残基。可以分别输入对应组别编号或名称。之后软件会计算两组之间的距离轨迹,输出一个包含时间点和对应距离的文本文件,方便后续绘图及分析。 在实际应用中,常见的操作流程包括:先使用make_ndx选择或构建目标残基的原子组;随后利用gmx distance计算两组目标之间的距离;最后利用数据可视化工具(如Grace、Matplotlib)绘制距离随时间变化曲线,观察结构变化趋势。 此外,可结合gmx mindist工具检测两组目标间的最短距离,或使用gmx pairdist计算特定原子对之间的距离分布。根据研究需求,还可调整采样时间、原子选择方式及轨迹解析设置,提升分析结果的精准度。
计算残基间距离时要注意轨迹文件是否已进行周期条件处理,如去除分子穿越边界的现象(使用trjconv命令加上-pbc nojump参数)。这一步骤非常重要,避免距离计算因人为周期性边界条件而产生误差。 在蛋白质结构动态研究中,残基间距变化可反映二级结构的稳定性或转变,辅助理解折叠过程。对配体绑定位点附近残基的距离分析有助于推断结合机制和亲和力变化,指导药物设计。 利用GROMACS计算残基间距离的灵活性允许用户深入探究多种生物大分子和复合体系,包括蛋白质-蛋白质、蛋白质-DNA等相互作用。丰富的命令工具组合使得残基距离测量准确且高效,成为分子模拟分析中不可或缺的技术。
总结来看,掌握GROMACS中计算残基间距离的方法不仅能揭示分子结构的动态信息,还能为功能解析和新药研发提供重要数据支持。用户在操作时应重视索引文件的合理划分、轨迹预处理和数据后期分析,以确保结果的科学性和可靠性。通过不断实践和深入理解,科研人员可以充分发挥GROMACS的强大功能,推动生命科学领域的创新研究发展。 。