在生物信息学领域,解析蛋白质结构和功能之间的关系一直是研究的热点之一。蛋白质的三维结构决定了其生物功能,而结构中氨基酸之间的接触信息对于理解蛋白质交互作用和设计新药至关重要。因此,准确预测蛋白质的接触图成为计算生物学的重要任务。近年来,多序列比对(Multiple Sequence Alignment,简称MSA)的统计模型,尤其是基于Potts模型的伪似然估计方法(plmDCA),在蛋白质接触预测中表现出强大的效果。Evorca作为一种利用JAX实现的快速且轻量级的plmDCA工具,提供了一个高效且易于使用的解决方案。本文将深入探讨Evorca的设计理念、功能特点及其在科研应用中的价值。
Evorca的核心优势在于它基于JAX和Optax两个现代化的深度学习框架,充分利用自动微分和高性能计算的优势,实现了plmDCA算法的加速。JAX通过将NumPy代码转换为高效的GPU或CPU代码,使得大规模多序列比对数据的处理和训练过程得以极大优化。结合Optax的AdamW优化器,Evorca能在保持模型准确性的同时,显著提升训练速度和资源利用效率。 此外,Evorca非常注重工具链的轻量化和用户体验。它提供了命令行接口(CLI)和Python API两种使用方式。研究人员既可以通过CLI轻松完成从MSA数据拟合到接触图可视化的全过程,也可以通过Python接口灵活调用各个功能,结合自身分析流程进行定制化开发。
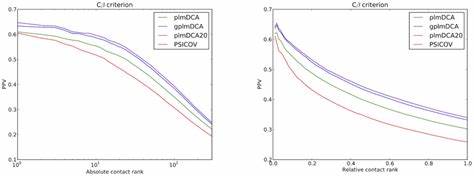
Evorca支持处理蛋白质和RNA两种序列类型,自动识别和清理插入,转换成适合Potts模型的稀疏张量表示。 在多序列比对处理方面,Evorca采用Henikoff风格的序列加权方法来减少同源序列冗余带来的偏差,该方法忽略gap位点,确保计算的稳定性和准确性。训练阶段采用负伪似然函数作为目标函数,并对单体参数及协变量参数施加L2正则化,以防止过拟合和参数发散。训练过程中的重要操作包括对称化耦合矩阵、去除自环影响以及施加零和值规范,使得最终输出的耦合矩阵更具生物物理意义。 预测结果以Frobenius范数衡量耦合参数的强度,从而映射成蛋白质接触概率矩阵。Evorca还可选地应用平均乘积校正(APC)方法,进一步优化接触预测的准确度和特异性。
结果可视化功能将高得分区域绘制成联系图,方便科研人员直观理解蛋白质结构的空间依赖关系。 Evorca的安装和使用门槛较低。只需通过pip命令安装核心库,便可快速开始分析。对于需要GPU加速的用户,只要按照JAX官方说明安装相应版本,即可享受显著的性能提升。文档中提供全面的使用范例,包括如何从A3M格式的MSA文件拟合模型,如何生成接触图,以及如何调用Python API进行自定义分析。这极大地方便了初学者和资深研究员的不同需求。
Evorca不仅是一款性能强劲的工具,也是开放源代码项目,用户可以根据自身科研需求自由拓展和改进。借助JAX的灵活性,研究者能够简单地调整模型细节或优化策略,探索更加复杂的统计模型。这为生物物理学、结构生物学等领域的交叉研究带来了极大的便利。 在科学研究中,准确的接触预测能够辅助蛋白质折叠模拟、变异效应分析和药物设计。Evorca通过高效地挖掘多序列比对中的共演化信息,为这些应用提供了坚实的技术基础。同时,其开源和模块化设计降低了学习与使用的难度,有助于推动相关领域的技术普及和创新。
展望未来,随着计算资源的不断升级和深度学习方法的结合,类似Evorca这样的工具将继续进化,提供更加精准和快速的分析能力。结合结构预测的最新进展,如AlphaFold等,plmDCA模型在多组学数据整合和功能预测中也将发挥更大作用。Evorca作为该领域内的创新尝试,展示了JAX生态系统中生物信息学工具开发的巨大潜力。 总之,Evorca通过轻量级、高性能的设计理念,赋能科研人员快速构建和训练Potts模型,实现多序列比对的深度统计分析及蛋白质接触预测。它不仅简化了复杂算法的应用流程,还确保了结果的科学性和可靠性。在现代计算生物学中,Evorca为结构预测和蛋白质功能解析提供了强有力的工具支持,值得广大科研人员关注和使用。
。